机器学习输入数据应该如何标准化
引言
效果展示
过程
选用指引
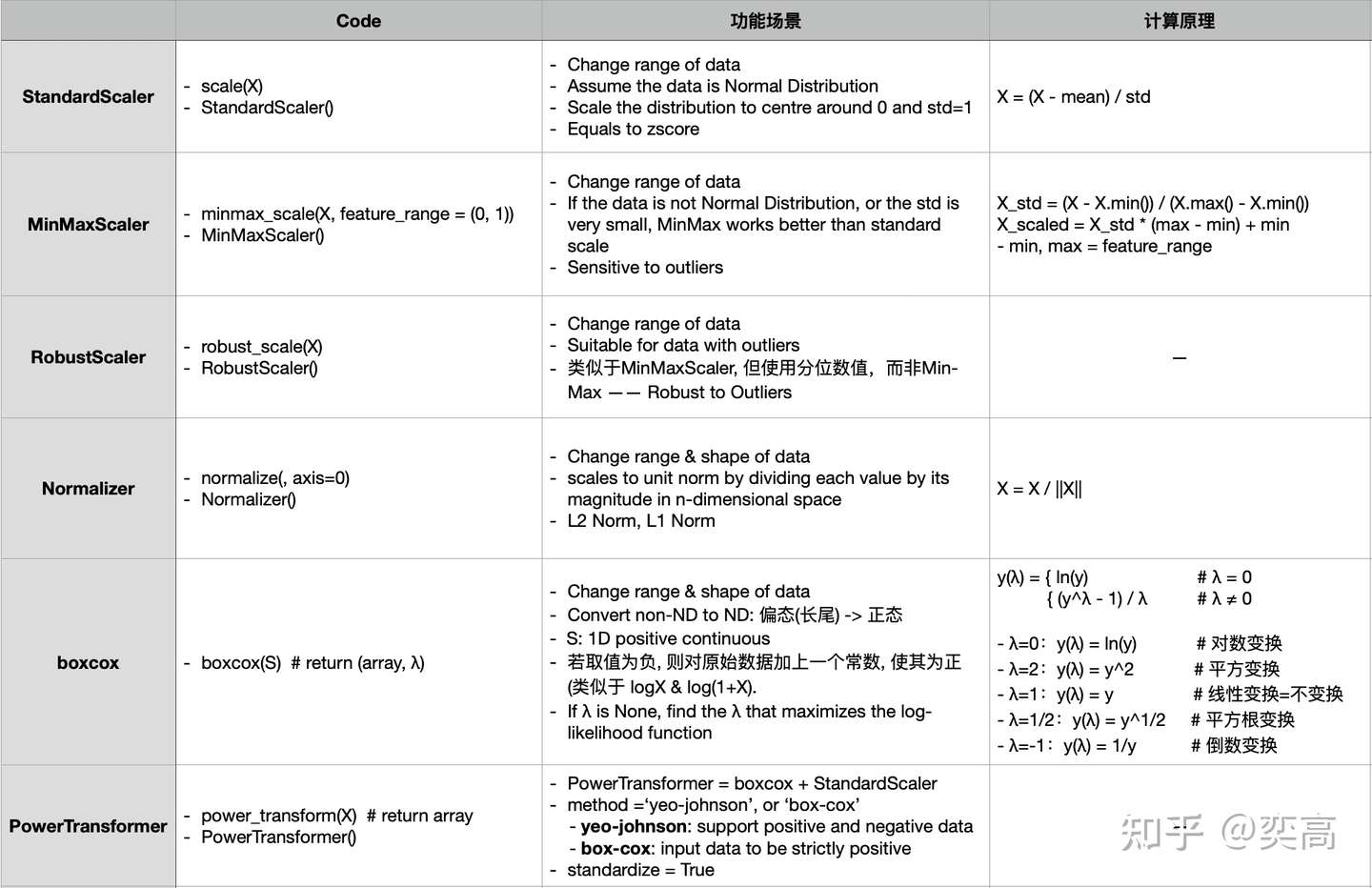
机器学习数据标准化的六种方法[1]
1 | if data == 'Non-Normal Distribution': |
判断是否满足正态分布的方法[2,3,4,5]
是否归一化
在机器学习中,基因表达量通常使用测序数据的读取计数或归一化读取计数作为值。这些值可以通过 RNA 测序(RNA-seq)获得,这是一种使用高通量测序技术来识别和定量转录 RNA 的方法。然后,这些读取计数可以进一步归一化以消除实验偏差和技术偏差。
例如,常见的归一化方法包括 TPM(每百万转录本)和 FPKM(每千基因每百万映射)或 RPKM(每千基因每百万映射)。这些方法考虑了基因长度和测序深度的影响,使得不同样本之间的基因表达量可以进行比较。
然后,这些值可以用作机器学习模型的输入特征,例如用于预测疾病状态或疾病亚型。然而,由于基因表达数据通常具有高维度、噪声多和样本少的特点,因此在使用机器学习模型处理基因表达数据时,需要采取适当的特征选择和模型选择策略。此外,还需要进行适当的交叉验证来评估模型的性能。
在机器学习中,基因表达量通常使用测序数据的读取计数或归一化读取计数作为值。这些值可以通过 RNA 测序(RNA-seq)获得,这是一种使用高通量测序技术来识别和定量转录 RNA 的方法。然后,这些读取计数可以进一步归一化以消除实验偏差和技术偏差。
例如,常见的归一化方法包括 TPM(每百万转录本)和 FPKM(每千基因每百万映射)或 RPKM(每千基因每百万映射)。这些方法考虑了基因长度和测序深度的影响,使得不同样本之间的基因表达量可以进行比较。
然后,这些值可以用作机器学习模型的输入特征,例如用于预测疾病状态或疾病亚型。然而,由于基因表达数据通常具有高维度、噪声多和样本少的特点,因此在使用机器学习模型处理基因表达数据时,需要采取适当的特征选择和模型选择策略。此外,还需要进行适当的交叉验证来评估模型的性能。
结论
引用
- 机器学习数据标准化的六种方法 - 知乎
- Python 篇 | 正态性检验方法详解 - 知乎
- 用 Python 检验数据正态分布的几种方法 - 知乎
- scipy.stats.normaltest — SciPy v0.14.0 Reference Guide
- 二元对数正态分布 (bivariate lognormal distribution) 的几个性质_bivariate normal-CSDN 博客
- Python Scipy stats.normaltest() 用法及代码示例 - 纯净天空
- 【答疑解惑-II】——不满足正态分布的数据到底能不能用 Gaussian process 的方法呢? - 知乎

