使用 nbia-data-retriever 在 linux 下载数据
引言
美国癌症医学影像档案中心 (The Cancer Imaging Archive, TCIA) 可以下载癌症病例数据,对于 mac 和 win 来说下载软件使用就可以了,但是在连接 linux 的 terminal 可能会出现两个报错:
一个是zsh: command not found: nbia-data-retriever, 另一个是java.awt.HeadlessException: No X11 DISPLAY variable was set。
第一个意味着系统无法在它查找可执行文件的标准路径(即 $PATH 环境变量指定的目录)中找到名为 nbia-data-retriever 的可执行文件。
第二个表明 nbia-data-retriever 这个程序(它是一个 Java 应用程序)试图执行一个需要图形界面(GUI)的操作,但在您当前运行的环境中没有找到可用的显示设备(X11 DISPLAY)。简单来说,这个程序可能在启动时尝试弹出一个窗口(比如用户协议、进度条或配置界面),但您正在一个没有图形界面的终端会话中运行它(这在服务器上很常见)。
1 | tenney@sxmu-PowerEdge-R750 $ sudo -S dpkg -i nbia-data-retriever_4.4.3-1_amd64.deb |
本教程所用的平台是 ubuntu 22.04 + macos 15.3.2.
效果展示
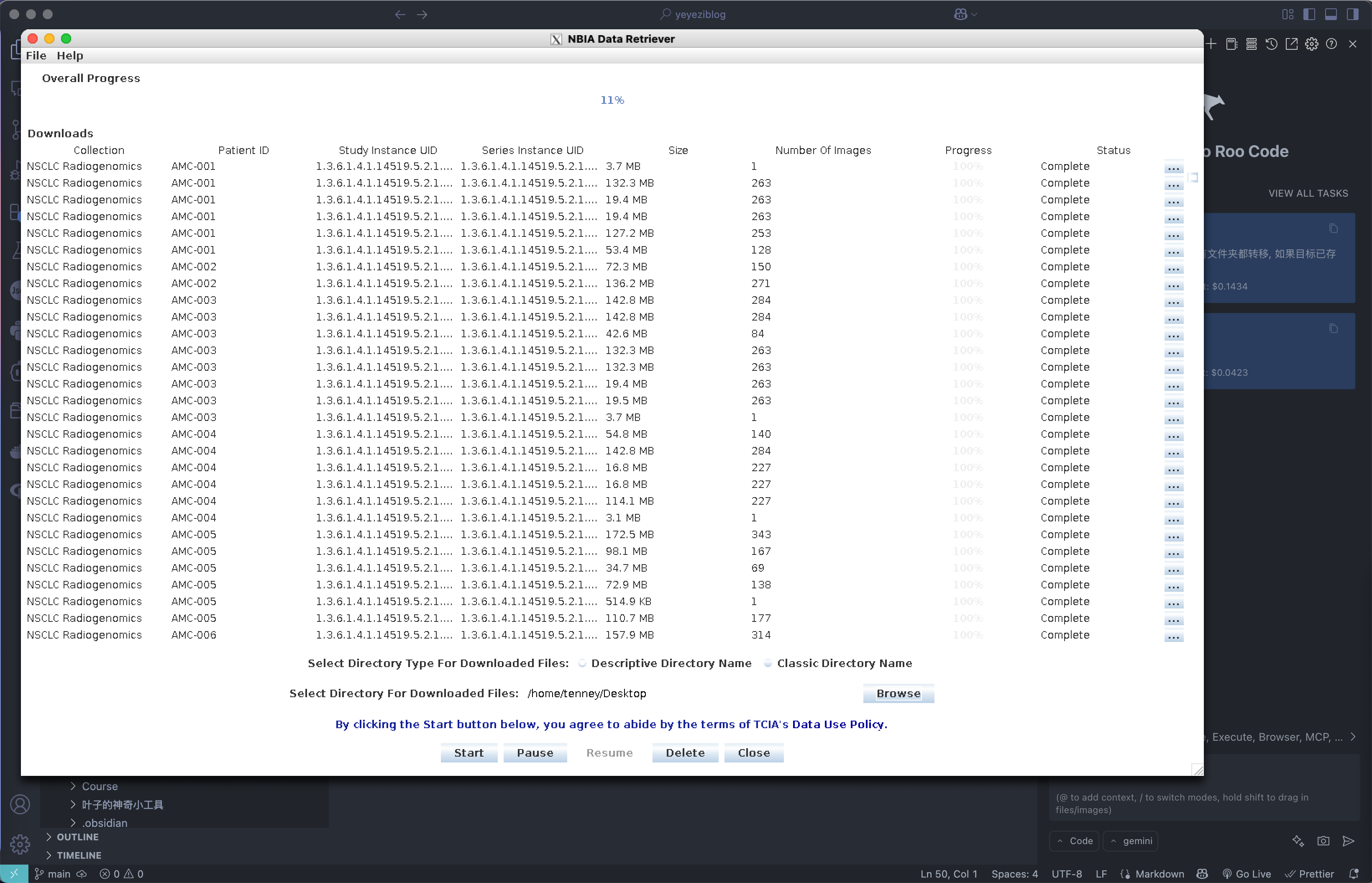
过程
安装 nbia-data-retriever
官网提供了安装指南[1].
对于 ubuntu 使用 deb 安装包安装,相应的 contos 使用 rpm.
1 | wget https://github.com/CBIIT/NBIA-TCIA/releases/download/DR-4_4_3-TCIA-20240916-1/nbia-data-retriever_4.4.3-1_amd64.deb |
但是安装后想使用就会出现引言里面出现的问题,解决方法我使用的是找到路径别然后直接执行,而不是加入 PATH.因为 PATH 里面已经塞了太多没必要的东西了完全没必要…
另外就是没有 conda 安装方式比较可惜,在 anaconda 官网也没搜到。
1 | dpkg -L nbia-data-retriever |
所以可执行目录就是/opt/nbia-data-retriever/bin/nbia-data-retriever.
下载数据
以https://www.cancerimagingarchive.net/collection/nsclc-radiogenomics/数据为例。
1 | wget https://www.cancerimagingarchive.net/wp-content/uploads/NSCLC_Radiogenomics-6-1-21-Version-4.tcia |
既然没有 X11 屏幕就用本地的好了,以前的教程VScode 使用 remote-ssh 实现服务器上绘图可视化 | Yezi’s Hexo Blog讲过如何配合 vscode 使用 r 绘图。跟那个类似,不过经过常年的锻炼我发现不用按照 r-studio 的思维走…
其实直接 terminal 里面跑就足足够够的。
服务端
如果是 centOS 可以使用yum install -y xauth xclock.
如果是 ubuntu 可以使用apt install -y xauth x11-apps.
本地准备
mac:
首先安装 xquartz 用来展示。
1 | brew install xquartz |
然后修改本地的./.ssh/config文件内容,加上ForwardX11 yes等三行。
1 | Host 32_local |
1 | ssh -Y tenney@10.9.65.32 |
-Y参数可以启用X11 Forwarding功能。
这里面有一个坑,就是 vscode 的remote X11可能是太久没更新已经不支持了,我在 vscode 里面无法调用到 DISPLAY/X11, 但是 terminal 可以,所以建议直接用 terminal.
如果成功输出了类似上面localhost:11.0的东西,就可以重跑刚刚的代码了。
Windows 听说是用 Xming.
结论
计算机是魔法,这个发明工具远远快于知道工具的速度的年代,就不要对工具刨根问底了,能用就行…
